Coordenadas tortuga y GSL
Coordenadas tortuga y GSL
Por cuestiones que aún no puedo desvelar (misterio misterioso) estoy necesitando obtener las coordenadas tortuga, r(r *), de forma numérica. Por lo visto para el estudio de Bounce Black holes y Wormholes es relativamente fácil obtener r *( r) de forma analítica pero muy difícil obtener r(r *).
Para entrenar un poco antes de ir al ring podemos estudiar el caso de Schwarzschild.
Schwarzschild
En el caso de Schwarzschild sabemos que r *( r) = (1 - rS/r)^-1 y por tanto r(r *) = (1 - rS/r), o sea, que tenemos ambas expresiones analíticas lo que es perfecto para probar nuestros métodos.
GSL
Voy a esbozar aquí una solución sin el uso de librerías de precisión variable y con memoria estática pero el uso de una librería como GMP te va a ser imprescindible si quieres llegar a la región asintótica del comportamiento de tu agujero negro. La memoria dinámica es prescindible… pero te permite experimentos ‘absurdos’ con por ejemplo 10 millones de subintervalos (que puede parecer mucho pero que GSL y GMP se meriendan en un abrir y cerrar de ojos) en tu intervalo de estudio (que orientativamente debe estar entre [-150, 150]).
La nomenclartura que estoy utilizando es:
- r = x
- r * = y
#define SYS_DIM 1
#define NODES 100001
#define SCH_PLU_INF 100000
#define SCH_MIN_INF -SCH_PLU_INF
/* Integration method */
#define FORWARD_INTEGRATION 0
#define BACKWARD_INTEGRATION 1
int m_integration = BACKWARD_INTEGRATION;
struct yxParams
{
double rS; /* Schwarzschild radius */
};
double m_rS = 2;
double yxAnalytical[NODES];
double yxCalculated[NODES];
/**
@brief yx[] Jacobian: -rS/(x^2*(rS/x - 1)^2)
*/
int
jacoYX(double t,
const double y[],
double *dfdy,
double dfdt[],
void *params)
{
(void)(t);
struct yxParams *par = (struct yxParams*)params;
double rS = (par->rS);
double x = y[0];
gsl_matrix_view dfdy_mat = gsl_matrix_view_array(dfdy, 1, 1);
gsl_matrix *m = &dfdy_mat.matrix;
gsl_matrix_set (m, 0, 0, -rS/(x*x*(rS/x - 1)*(rS/x - 1)));
dfdt[0] = 0.0;
return GSL_SUCCESS;
}
/**
@brief ODE IV 1st grade to solve y[x]'
@param t Independent variable
@param y[] Left side of the 1st grade system of equations
@param sys[] Right side of the 1st grade system of equations
*/
int
funcYX(double t,
const double y[],
double sys[],
void* params)
{
struct yxParams *par = (struct yxParams*)params;
double rS = (par->rS);
(void)(t);
double x0 = y[0];
assert(x0 != 0);
sys[0] = 1.0/(1.0 - rS/x0);
return GSL_SUCCESS;
}
/**
@brief r *( r) integration... so the inverse r(r *) tortoise coordinates.
We do this because it's easier to get an analytic expression from
r *( r) that from r(r *)
@param IN, a: interval lower limit
@param IN, b: interval upper limit
@param IN, n: number of subintervals
@param IN, ic: Initial condition
@param OUT, yx[]: Array with the integration values
*/
int
yx_integration(double a, double b, int n, double iC)
{
int status = GSL_SUCCESS;
double rS = m_rS;
double h = (b - a)/n;
double x0 = a;
double x1 = x0 + h;
double epsAbs = 0;
double epsRel = 1e-6;
struct yxParams params = {rS};
gsl_odeiv2_system sys = {funcYX, jacoYX, SYS_DIM, ¶ms};
const gsl_odeiv2_step_type* T = gsl_odeiv2_step_msbdf; /* rkf45 rk8pd , rk4imp bsimp msadams msbdf*/
gsl_odeiv2_driver * d = gsl_odeiv2_driver_alloc_y_new (&sys, T, h, epsAbs, epsRel);
double y[1] = {iC};
int i;
for (i = 0; i < NODES; i++)
{
status = gsl_odeiv2_driver_apply(d, &x0, x1, y);
x0 = x1;
x1 = x0 + h;
if (status != GSL_SUCCESS)
{
printf ("error, return value = %d\n", status);
printf("x = %f, y = %f\n", x0, y[0]);
break;
}
else
{
yxCalculated[i] = y[0];
}
}
gsl_odeiv2_driver_free(d);
return status;
}
/**
@brief Tortoise analytical expression: r *( r)
@param IN a: interval lower limit
@param IN h: step
*/
void
yx_analytical(double a, double h)
{
double rS = m_rS;
double x = a;
int i;
for(i = 0; i < NODES; i++)
{
double tortoise = x + rS*log(x/rS - 1);
if(_isnanf(tortoise))
{
tortoise = SCH_MIN_INF;
}
yxAnalytical[i] = tortoise;
x += h;
}
}
/**
@brief Save the data so we can plot it
@param IN x
@param IN h
*/
void
txt_data(double x, double h)
{
FILE *fp;
fp = fopen("data.txt", "w");
int i;
for (i = 0; i < NODES; i++)
{
if(m_integration == FORWARD_INTEGRATION)
fprintf(fp, "%.32f %.32f %.32f %.32f\n", yxAnalytical[i], x, yxCalculated[i], x);
else
fprintf(fp, "%.32f %.32f %.32f %.32f\n", yxAnalytical[i], x, yxCalculated[NODES - i], x);
x += h;
}
fclose(fp);
}
/**-----------------------------------------------------------------------------
------------------------------------------------------------------------------*/
int main()
{
/*
To our purposes these values are enough far away from the BH.
These values are for the numerical integration.
*/
double b = 150;
double a;
if(m_integration == BACKWARD_INTEGRATION)
a = -b;
else if(m_integration == FORWARD_INTEGRATION)
a = m_rS + 1e-6;
int n = NODES - 1;
double h = (b - a)/n;
/* Do calculations... */
yx_analytical(a, h);
if(m_integration == BACKWARD_INTEGRATION)
{
double ic = b + m_rS*log(b/m_rS - 1);
yx_integration(b, a, n, ic);
}
else
yx_integration(a, b, n, a);
/* Save the data in a text file */
txt_data(a, h);
return 0;
}Podemos integrar hacia delante o hacia atrás. Si lo hacemos hacia delante lo haremos desde el horizonte + delta hasta +inf. 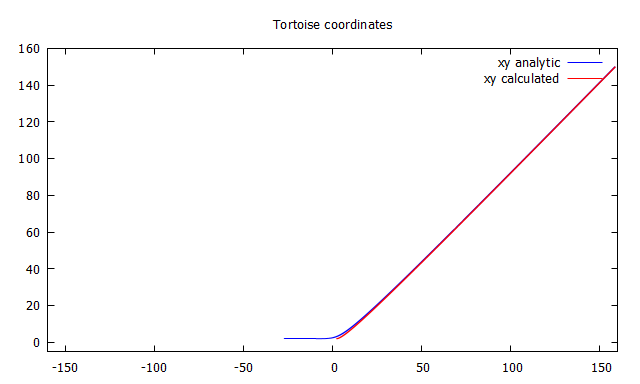
Coordenada tortuga integrando hacia delante
También puedes integrar hacia atrás… y aquí es donde nos encontramos la sorpresita. Resulta que al hacer la integración hacia atrás, partimos de la aproximación que r * = r para ‘r’ grandes… pero no nos vale porque obtenemos la gráfica correcta pero con un ‘offset’ 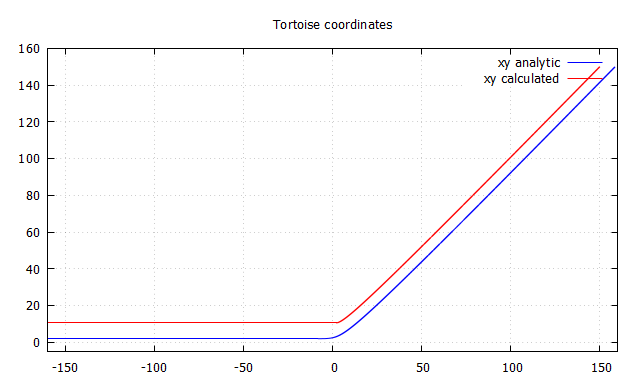
Coordenada tortuga integrando hacia atrás offset
Veamos un ejemplo numérico (de esos que no gustan nada a los físicos XDDD). Para nuestro infinito 150 obtenermos una r * = 158 (aprox). Es decir 8 de offset. Para r = 100.000 tenemos r = 100.021 (hablo de memoria) es decir 21 de offset… pero en realidad la relación 100.000 vs 100.021 es muchísmo menor que 150 vs 158. Es decir, efectivamente r * = r para r grande aun cuando la diferencia entre el valor exacto y el valor aproximado es mayor.
Para trabajar con esta burrada de intervalos y valores necesitamos utilizar memoria dinámica. Como digo GSL se lo merienda rápido rápido.
Entoces ¿qué hacemos para eliminar ese offset? pues la única solución es utilizar como condición inicial el valor exacto. 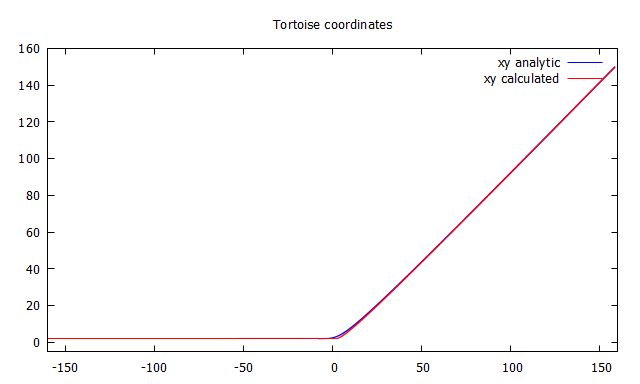
Coordenada tortuga integrando hacia atrás
Lo bueno del método de integración hacia atrás es que te olvidas de saber si hay un punto asintótico, dónde está, etc. Pero a cambio te impone conocer el valor exacto de la condición inicial.
Y sí, efectivamente si no conoces la condición inicial exacta obtienes la forma de la gráfica correcta… pero desplazada
¿Cómo dibujas r(r *) si hemos obtenido r *( r)?
Pues simplemente tienes que intercambiar x <–> y como ves en las líneas
...
if(m_integration == FORWARD_INTEGRATION)
fprintf(fp, "%.32f %.32f %.32f %.32f\n", yxAnalytical[i], x, yxCalculated[i], x);
else
fprintf(fp, "%.32f %.32f %.32f %.32f\n", yxAnalytical[i], x, yxCalculated[NODES - i], x);
...Pero en realidad lo ‘más destacable’ es que al haber calculado la jacobiana, puedo utilizar tanto métodos explícitos como métodos implícitos (de hecho aquí estoy usando un método implícito)
...
const gsl_odeiv2_step_type* T = gsl_odeiv2_step_msbdf; /* rkf45 rk8pd , rk4imp bsimp msadams msbdf*/
...Pues aquí va otro ejemplo de uso de la fantástica librería GSL que no es el típico Var der Pol.